プレスリリース
国立研究開発法人海洋研究開発機構
情報・システム研究機構 国立遺伝学研究所
海洋細菌叢・ウイルス叢が持つDNA化学修飾(エピゲノム)を大規模に解明
1. 発表のポイント
- ◆
- 海洋微生物叢を対象に「メタエピゲノム解析」を実施し、ゲノム上のDNA化学修飾(エピゲノム)を大規模に解明した。
- ◆
- 新規のDNA化学修飾酵素を複数同定したほか、エピゲノムは微生物の進化や生態などに広範な影響を与えている可能性が示唆された。
- ◆
- 今後、海洋のみならず土壌や腸内といった幅広い環境に生息する微生物叢を対象に解析を実施することで、従来のゲノム解析を越えた、より深い微生物生態系や進化の理解が進むことが期待される。
2. 概要
国立研究開発法人海洋研究開発機構(理事長 松永 是)海洋機能利用部門 生命理工学センター 深海バイオリソース研究グループの平岡 聡史 研究員らは、情報・システム研究機構 国立遺伝学研究所と共同で、房総半島沖から採取された海水試料を分析し、海洋微生物叢が持つゲノム上のDNA化学修飾(エピゲノム)を大規模に明らかにしました。
細菌や古細菌、二本鎖DNAウイルスは、ヒトなどの真核生物と同様に、生体内でゲノムDNAに化学修飾が起きることが知られており、生理学的に重要な役割を担うと考えられています。そのため、微生物のゲノム情報や遺伝子情報とあわせてエピゲノム情報を取得し、その機能を明らかにしていくことは、海洋や土壌、腸内といったさまざまな環境に生息する微生物の生理生態を理解する上で重要です。しかしながら、DNA化学修飾を観測する技術的な難しさや、環境中の微生物叢の大半を占める未培養系統が存在するため、微生物エピゲノムに関する研究は十分に進んでいません。
そこで本研究では、メタゲノム解析とエピゲノム解析を組み合わせた「メタエピゲノム解析」 と呼ばれる非培養的な解析手法を利用して、表層から深海に至る4層の海水試料を対象に、微生物叢が持つDNA化学修飾をより大規模に解析しました。その結果、海洋微生物叢が持つエピゲノムの多様性や系統的分布が、初めて明らかになりました。さらに、これらのDNA化学修飾を引き起こす新規酵素を複数発見したほか、より詳細なゲノム解析からDNA化学修飾が微生物の進化や生態に密接に関わることが示唆されました。本研究は、塩基配列のみを解析対象とする狭義のゲノム解析を越えて、DNA化学修飾(エピゲノム)をも含む広義のゲノム解析を行うことで、微生物の生理生態や進化により深くアプローチできることを示した成果です。
本成果は「Nucleic Acids Research」に1月21日付でオンライン公開される予定です(オープンアクセス)。なお、本研究の一部は文部科学省 科学研究費補助金(JP16H06429、JP16K21723、JP16H06437、JP19H05684、JP19H05679)、日本学術振興会 科学研究費補助金(P18K11636、JP19H04246、JP19K21203、JP20K15444、JP20H02020)、および公益財団法人発酵研究所 若手研究者助成(Y-2020-2-017)によって実施されました。
(DOI: 10.1093/nar/gkab1292)
- 国立研究開発法人海洋研究開発機構
- 国立遺伝学研究所
3. 背景
生命の設計図であるゲノムは、ATCGの4種類の塩基が鎖状に並んだDNAと呼ばれる分子から作られています(※1)。そして、ゲノムには酵素の働きにより、さまざまな種類の化学修飾がアクセサリーのように後天的に施されることがあり、この仕組みは「エピゲノム」と呼ばれています(※2)。このようなゲノムへの化学修飾は、遺伝子の発現制御や幹細胞の分化、細胞の癌化、疾患の発症など、多様な生命現象に関わる機構の一つとして知られており、ヒトやマウスをはじめとした真核生物を対象に、精力的に研究が進められています。
一方で、細菌や古細菌といった原核生物、そして二本鎖DNAウイルスにおいても、ゲノムDNAへの化学修飾が起きることが昔から知られており、真核生物と同様に生理学的に重要な役割を果たしていると考えられています。実際にさまざまな微生物系統において、遺伝子発現調節やウイルス感染に対する防衛機構(制限修飾系)、DNA修復、細胞周期の制御など、多彩な機能に関わる例が報告されています(図1)。
しかしながら、原核生物やDNAウイルスが持つDNA化学修飾の実験的な検出には技術的な制約があり、真核生物と比較して研究例が圧倒的に少ない現状があります。また、海洋をはじめとする自然環境中の微生物の大半は、実験室で培養することができない、いわゆる未培養系統に属していると考えられていますが、今日までの微生物エピゲノム研究の多くは、実験室で培養が容易なごく一握りの系統のみを研究対象としてきています。そのため、微生物エピゲノムの生態学的・進化学的な意義はもとより、未培養系統を多く含む地球上の微生物全体においてどのようなエピゲノムの多様性が存在するのか、といった基礎的なことすら十分にはわかっておらず、DNA化学修飾が持つ機能や与える影響を解明する上で障害となっています。
これまでに、本発表論文の筆頭著者を中心とした研究グループでは、PacBio社製シーケンサーを活用し、メタゲノム解析とエピゲノム解析を組み合わせることで、微生物叢が持つエピゲノムを非培養的に解析する手法「メタエピゲノム解析」を世界に先駆けて確立し、その有効性を実証しました(2019年1月17日付東京大学プレスリリースを参照)。本研究では、PacBioに加えてOxford Nanopore社製シーケンサーやIllumina社製シーケンサーを併用し、今までエピゲノム解析の対象とされてこなかった海洋微生物叢を対象とした、大規模なメタエピゲノム解析を実施しました。
4. 成果
2019年9月、海底広域研究船「かいめい」を用いて房総半島沖合50 kmの海域から大量採水を行い、フィルター濾過により表層5 mから深層300mに至る4層の微生物サンプルを採取しました(図2)。これらのサンプルを対象に、PacBio Sequel、Nanopore GridION、Illumina MiSeqの3種類のシーケンサーを用いたショットガンシーケンスを行い、配列データを取得しました(図3)。そして、さまざまなバイオインフォマティクス解析を経て、233の原核生物のドラフトゲノム(P-MAG)と163の二本鎖DNAウイルスのドラフトゲノム(V-MAG)を取得しました。
PacBioのリードを利用してこれらのゲノム中のDNA化学修飾を検出し、その修飾モチーフ(※3)を予測したところ、新規のものを数多く含む、計220の多様な修飾モチーフを発見しました(図4)。原核生物ゲノムからは興味深いことに、系統的に近しいゲノム間でも大きく異なるエピゲノムを持つ場合もあれば、比較的遠い系統内でも高度に類似したエピゲノムを持つ系統群、DNA化学修飾が全く検出されない系統群など、さまざまなパターンが観察されました。また、ウイルスゲノムではDNA化学修飾が観察された系統群(Myoviridae科など)とそうでない系統群が明確に分かれていました。これらの結果は、それぞれの微生物系統において異なるエピゲノムの役割が存在し、生理生態と密接にリンクしていることを示唆しています。
次に、DNAメチル化と呼ばれるDNA化学修飾に特に着目して解析したところ、この種類の化学修飾を施す酵素として、計276のDNAメチル化酵素(MTase)遺伝子が予測されました。そして人工遺伝子合成と大腸菌を用いた組み換え実験、分離精製した酵素を用いた実験などから、5つの新規モチーフを特異的に認識する酵素を含む、計11のMTase酵素を新規に同定することに成功しました。
さらに、海洋中に最も多く存在する微生物系統群であるAlphaproteobacteria綱に着目してより詳細なゲノム解析を行ったところ、この系統群では進化の途中でDNAメチル化酵素の認識モチーフが変化し、その変化にあわせてゲノム全体の塩基出現パターンも変化していることがわかりました。このことは、エピゲノムとゲノムが共進化してきたことを示唆する、興味深い結果です。すなわちDNA化学修飾は、ゲノムに装飾的に付くアクセサリーのような存在でありながら、ゲノムそのものに大きな影響を与え微生物の進化を駆動する重要な因子の1つになり得る、という可能性が示唆されました。
5. 今後の展望
本研究は、今日まで解析対象とされてこなかった海洋細菌叢・ウイルス叢が持つエピゲノムを大規模に解析し、これまで知られていなかった多様なDNA化学修飾モチーフの存在を明らかにしたとともに、エピゲノムが海洋微生物の生態や進化に広範な影響を与えている可能性を示した初の成果です。
近年、ヒトを始めとする真核生物のエピゲノム研究が、特に医学的な側面から世界的に進められており、日々新たな発見が報告され数多くの論文が出版されています。一方で微生物については、さまざまな技術的制約も相まって、長年に渡りエピゲノム研究が滞っている状況が続いていました。しかしながら、PacBioシーケンサーの登場と発展により、分離培養株を用いた微生物のエピゲノムが簡単に計測できるようになり、ここ数年で状況は大きく変わりつつあります。さらに、本研究で用いたメタエピゲノム解析手法を用いることで、分離培養株のみならず自然界に存在する幅広い微生物系統についても、微生物エピゲノムの非培養的な解析が可能になっています。
このメタエピゲノム解析は、海水環境のみならず、幅広い微生物叢サンプルに適用可能な手法です。今後、今回とは異なる海域の海洋水や深海堆積物など、より多様な海洋環境を対象に解析を実施することで、海洋微生物が持つエピゲノムをより広範に解明できると考えられます。さらに海洋のみならず、土壌や腸内環境、温泉などの幅広い環境を対象に解析を実施し知見を積み重ねていくことで、微生物が持つエピゲノムへの理解が飛躍的に深まることが期待されます。
また、エピゲノムという微生物分野では従来あまり光を浴びてこなかった情報を活用することで、塩基配列という狭義のゲノム情報のみでは説明がつかなかった生命現象の解明が進む可能性も考えられます。エピゲノムが駆動する微生物生態や微生物進化の理解が進むことで、幅広い産業分野における微生物利用やバイオテクノロジー開発などにも発展的に繋がっていくことが期待されます。
【補足説明】
- ※1
- 例外として、コロナウイルスやインフルエンザウイルスなどのRNAウイルスでは、DNAではなくRNAと呼ばれる分子でゲノムが作られている。海洋にもさまざまなRNAウイルスが存在することが知られているが(2018年 9月7日既報)、本研究では二本鎖DNAを持つ生物(細菌、古細菌)とウイルス(二本鎖DNAウイルス)に焦点を絞って解析を実施した。
- ※2
- 真核生物のエピゲノムに関わる化学修飾にはさまざまな種類が知られており、例えば塩基に対するメチル化・アセチル化・ヒドロキシメチル化などのほか、DNAが巻き付いているヒストンタンパク質に対するメチル化・アセチル化・リン酸化・ユビキチン化、などが挙げられる。一方で原核生物やウイルスは、一般にヒストンタンパク質を持たないため、DNAへの直接的な化学修飾が主な研究対象となっている。
- ※3
- 原核生物やDNAウイルスでは、修飾酵素の働きによってDNAの塩基に化学修飾が施される。修飾酵素は一般的に、4から10塩基程度の長さからなる特定の塩基配列を認識し、その内の1塩基に対して化学修飾を施す。例えばM.EcoRIというDNAメチル化酵素は、GAATTCという並びを持つ塩基配列を認識し、下線が引かれた二番目のAに対してメチル基を修飾する。この場合、M.EcoRIはGAATTCをモチーフとして認識する、と表現される。
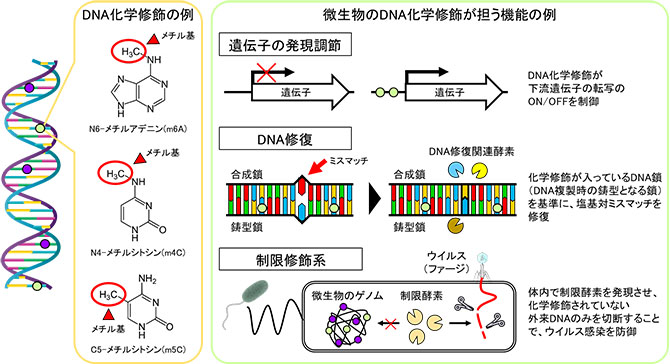
図1 微生物エピゲノムの概要。代表的なDNA化学修飾の例(左)と、DNA化学修飾が担っている機能の一般的な例(右)を示した。
※画像素材として、Togo Picture Gallery(© 2016 DBCLS TogoTV)を利用。
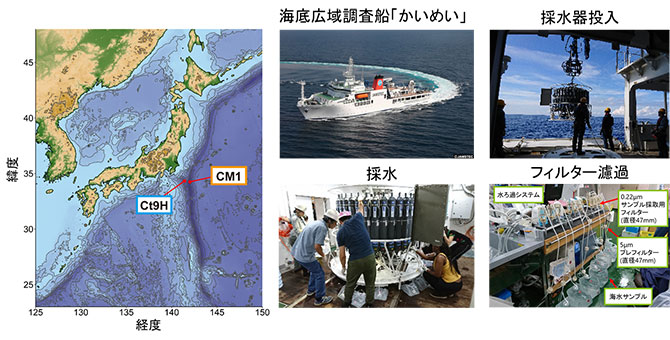
図2 サンプリング地点(左)とサンプリングの様子(右)。
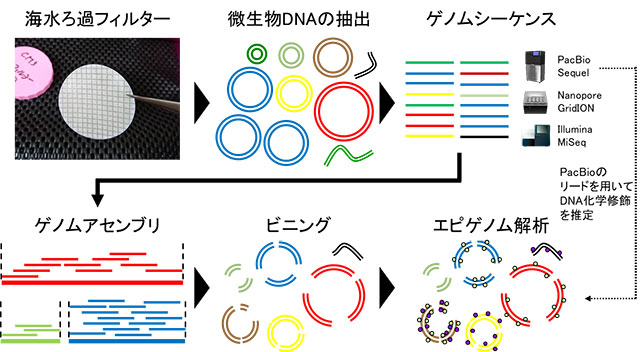
図3 解析の概要。最初に、海水ろ過によってフィルター上に捕集した微生物サンプルを用いて、DNA抽出とゲノムシーケンスを行う。次に、得られた大量の塩基配列情報を用いて、ゲノムアセンブリやビニングと呼ばれる情報解析を行い、もともとの微生物ゲノム配列を再構築する。得られたゲノム配列を利用して、PacBioリードとあわせて解析することで、DNA化学修飾のゲノム上の位置と種類を推定することができる。PacBioは、特にN6-メチルアデニン(m6A)とN4-メチルシトシン(m4C)という2種類のDNA化学修飾を高感度に検出することができる特徴がある。
※画像素材として、Togo Picture Gallery(© 2016 DBCLS TogoTV)を利用。
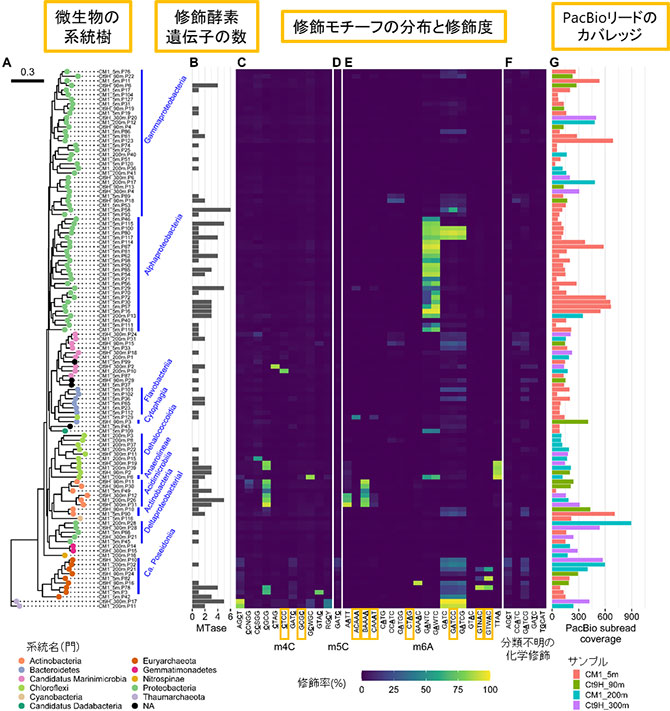
図4 海洋細菌・古細菌が持つエピゲノムの分布。(A)本解析で得られたドラフトゲノムに基づいて推定された、微生物の系統樹。ノードの色は、その微生物の系統名(門)を示している。 (B)ゲノム中から検出されたDNAメチル化酵素(MTase)遺伝子の数。(C-F)修飾モチーフの修飾度。それぞれのゲノムで、それぞれのDNA化学修飾モチーフがどれだけ修飾されているのかをヒートマップにより表現している。例えば明るい黄色のセルは、そのモチーフがその微生物のゲノム全域で高頻度に修飾を受けていることを示している。修飾モチーフは化学修飾の種類により分類しており、それぞれ(C)m4C(N4-メチルシトシン)、(D)m5C(C5-メチルシトシン)、(E)m6A(N6-メチルアデニン)、(F)修飾の種類を判別できなかったもの、を示している。また、今までに論文などで報告されておらず本研究で初めて発見されたと考えられるDNAメチル化修飾の修飾モチーフを黄色の枠線で示している。(G)PacBioより出力されたリード(subreads)のカバレッジ。バーの色は、それぞれのゲノムが得られたサンプルを示している。
- (本研究について)
- 国立研究開発法人海洋研究開発機構
- 海洋機能利用部門 生命理工学センター 深海バイオリソース研究グループ
研究員 平岡聡史 - (報道担当)
- 国立研究開発法人海洋研究開発機構
- 海洋科学技術戦略部 報道室
- 国立遺伝学研究所
- リサーチ・アドミニストレーター室 広報チーム