プレスリリース

国立研究開発法人海洋研究開発機構
国立大学法人筑波大学
海洋微生物の中に隠された新しいウイルスワールドを発見
~RNAウイルス網羅検出技術の開発と海洋微生物への適用~
1. 概要
国立研究開発法人海洋研究開発機構(理事長 平 朝彦、以下「JAMSTEC」という)海洋生命理工学研究開発センターの浦山俊一ポストドクトラル研究員(当時。現 筑波大学生命環境系助教)、布浦拓郎研究開発センター長代理らの研究グループは、RNAウイルスを網羅的に検出する方法(Fragmented and primer Ligated dsRNA Sequencing 、以下「FLDS」という)を開発し、非常に多様なRNAウイルスが海洋微生物へ感染・共存していることを発見しました。
一般に、ウイルスは宿主生物(※1)に感染・増殖後、宿主細胞を破壊して飛び出し、新たな宿主生物を求めるとされています(図1)。そのため、細胞を飛び出した環境中のウイルス粒子を対象に解析することで、その場におけるウイルスの多様性・生態の評価が試みられています。ウイルスは大きく3タイプ(DNAウイルス、 RNAウイルス、レトロウイルス)に分類されますが(※2)、特にDNAウイルスについては、海洋環境等に非常に多様なウイルスが存在することが明らかにされています(図2)。一方、RNAウイルスについては、解析手法が確立しておらず、自然環境中のRNAウイルスの多様性・生態は殆ど知られていませんでした。
今回、研究グループは、RNAウイルスゲノムを網羅的に解析する手法を確立し、海洋環境におけるRNAウイルスの多様性評価を試みました。その結果、10Lの海水から842種(ほぼすべて新種)のRNAウイルスを検出し、それらは既知のRNAウイルスファミリーの約半数を占めるとともに残り半数は全く新規な系統群に分類されることを明らかにしました(図3)。さらに、特定のRNAウイルス群について微生物内に存在するものと海水中を浮遊しているものを比較し、その多くは微生物細胞内でのみ検出されることを示しました(図4)。このことは、微生物細胞内からのみ検出されたウイルスは、細胞を破壊して細胞外に飛び出すことなく、宿主微生物と共存していることを示唆します。
従来、ウイルスのほとんどは病原体として捉えられてきましたが、近年の研究では病徴を示さない生物にもウイルスが感染しているという事例が報告されるようになりました。今回の研究結果は、自然界において、ウイルスの多くは宿主と穏やかな共存関係を結んでおり、少なくともRNAウイルスにおいては宿主に著しい負荷を及ぼす病原性ウイルスは特異な一群である、という可能性を強く示しています(図5)。
RNAウイルスを網羅的に検出するFLDSは、ウイルスの生態研究だけでなく、幅広い分野で新たな生物学・バイオテクノロジーの展開に資することが期待される技術です。JAMSTECでは関係機関とともに医学・公衆衛生や農学分野での適用可能性を探る研究も開始しています。
本研究は、独立行政法人日本学術振興会(JSPS)による科学研究費助成事業(JP26892031、JP16H06429、JP16K21723及びJP16H06437)の助成を受けて実施されたものです。
本成果は、科学誌「Molecular Ecology Resources」に9月7日付け(日本時間15時)で掲載される予定です。
タイトル:Unveiling the RNA virosphere associated with marine microorganisms
著者名:Syun-ichi Urayama1,2, Yoshihiro Takaki1,3,4, Shinro Nishi1,4, Yukari Yoshida-Takashima3, Shigeru Deguchi1, Ken Takai3, Takuro Nunoura1,4
所属:1.JAMSTEC海洋生命理工学研究開発センター、2. 筑波大学生命環境科学研究科、3.JAMSTEC深海・地殻内生物圏研究分野、4.JAMSTEC次世代海洋資源調査技術研究開発プロジェクトチーム
2.背景
一般に、ウイルスは病原体として認識されていますが、必ずしもウイルスの性質を正しく捉えているとは言えません。1900年頃、人類は動物や植物の病原体を探す中で、初めてウイルスを見いだしました。その後の100年余りの間に、人類は様々な病気の原因因子として多数のウイルスを発見しましたが、ウイルスを探す動機として、「病気・病徴」という現象が必要でした。逆に、病気を起こさないウイルスは探索するきっかけを与えてくれないことから、従来のウイルス探索では見つけにくい存在だったと言えます。一方、イネをはじめとする作物に対して明確な病気を起こさずにRNAウイルスが持続感染していることが知られるように、一見して異常がない身の回りの生物にも、実は共存型のウイルスが隠れていることが多々あります。また、海水や土壌等の環境においても、その環境に生息する微生物細胞数の少なくとも10-100倍にも及ぶウイルス粒子が存在することが明らかにされています。これら自然環境のウイルス粒子の多くは微生物へ感染し、その細胞内で生産されると推測されていますが、その様子を見ることはできません。近年、このように明確な感染現象が見えないウイルスやウイルス集団に対して網羅的な環境ゲノム解析を実施し、どのようなウイルスが存在するのかを明らかにする研究が盛んに実施されています。しかし、その多くの事例はDNAウイルスが対象とされており、RNAウイルスについては、技術上の課題があることから、一部の先行事例で試みられているのみでした。
そこで、研究グループはRNAウイルスについても網羅的な解析が可能な解析技術の開発を目指しました。特に着目したのは宿主細胞中に存在するRNAウイルス由来の長鎖二本鎖RNAです。これまでも長鎖二本鎖RNAは病徴を示さないウイルスの探索において、分子生物学的な指標として用いられており、イネ等の共存型ウイルスは、 長鎖二本鎖RNAを利用して検出されてきました。長鎖二本鎖RNAは健常な細胞には殆んど存在しませんが、細胞にRNAウイルスが感染するとそのゲノムや複製中間体としてウイルス由来の長鎖二本鎖RNAが細胞内に蓄積されます(※3)。つまり、生物試料から長鎖二本鎖RNAを検出することで、宿主生物に病徴があるかどうかに関わらず、細胞内に存在する一本鎖・二本鎖を問わずRNAウイルスの存在を知ることができるのです。しかし、長鎖二本鎖RNAの全長配列を簡便に決定する手法は、これまで存在していませんでした(※4)。
本研究では、新たに確立した①細胞由来の長鎖二本鎖RNAの効率的な全長解析技術を、②海洋微生物群集に適応し、更に③細胞外RNAウイルス粒子の多様性と比較検討することにより、新たなRNAウイルス生態像を可視化することに成功しました。
3.成果
宿主細胞内の“長鎖二本鎖RNA”を指標とした独自の網羅的RNAウイルス探索手法FLDSを確立しました。これは、生物試料から精製した長鎖二本鎖RNAを断片化した後にcDNAを合成して配列解読し、コンピュータ上で断片化前の二本鎖RNAを再構成する手法です。これにより、従来手法とは質的に異なる高品質なRNAウイルス(一本鎖・二本鎖RNAウイルス)ゲノム情報を得ることが可能となりました。また高品質なゲノム情報が得られることで、既存の手法ではウイルスであるかどうかわからなかったような新規性の高い遺伝子配列情報に関しても、高い確度でウイルス遺伝子であることを推定可能となりました(※5)。
この手法を用い、RNAウイルスの多様性に関する情報が極めて限られていた海洋微生物に感染するRNAウイルス叢を明らかにするため、北太平洋の5地点より濾過して得た微生物集団を対象にFLDSによる解析を実施しました。また、比較対象として同じ試料から得たウイルス粒子についても解析を行いました。
結果、全体では、既知のRNAウイルス53グループのうち、約半数の23グループに属すると思われるウイルス群を検出しました。その他にも、全く新しい新奇RNAウイルスグループに属すると予想されるゲノム配列も多数検出したことから、海洋環境、特にそこに存在する微生物の中には膨大な種類の未知RNAウイルスが潜んでいることが明らかとなりました。
さらに、ウイルス粒子画分のRNAウイルス配列と細胞内から得たRNAウイルス配列を比較検討し(図2)、特に二本鎖RNAウイルスについては、微生物細胞から検出される多様なウイルスが、ウイルス粒子画分(※6)からはほとんど検出されないことを示しました(図4)。このことは、細胞外へ出るRNAウイルスは海洋微生物集団に感染するウイルスのごく一部に限られていること、逆に細胞外へ出ないウイルス集団は、宿主微生物を傷つけることなく、共存・共生状態を維持していることを示唆します。
また、少なくともRNAウイルスを対象とした環境ゲノム解析を実施するにあたっては、ウイルス粒子を対象とするより、細胞内ウイルス核酸(長鎖二本鎖RNA)を対象とする方が、その環境に生息するRNAウイルス集団の概要把握において明らかに効率的・効果的であることを示します。
4.今後の展望
本研究では、未知種から構成されるRNAウイルス集団を効率良くゲノム解析する手法を確立し、海洋微生物に数多くの未知ウイルスが存在することを明らかにしました。この結果は、ウイルスの生態・進化を理解するには、病徴という指標に頼ることなく多様な環境における多様な生物を探索する必要があること、その探索には細胞外に放出されたウイルス粒子のみを対象とするのではなく、細胞中の二本鎖RNAを解析対象として実施することが最も効率的であることを示しました。今後、このような新しい視点に基づくRNAウイルス探索が行われることで、これまでほとんど見過ごされてきた“明確な病気を引き起こさない”RNAウイルスを含む新たなウイルス生態像や、ウイルス進化の道筋が明らかになってくることが期待されます。
また、本研究の結果は、少なくともRNAウイルスにおいては、宿主にダメージを与えるのではなく、共存・共生する戦略が優占している可能性を示しています。もし多くのウイルスが生物との共存・共生を選んでいるのであれば、そこで何が起きているのか、ウイルスは生物の生存に貢献しているのか等、その機能を明らかにすることで、新たな生物学・バイオテクノロジーの展開が期待されます。
効率的なウイルス探索技術は、ウイルスの生態研究という基礎研究だけでなく、公衆衛生、農林畜産業等における防疫においても不可欠な技術です。FLDSと命名した独自技術は、様々な生物試料に含まれるRNAウイルスを従来の手法と比較し、極めて効率的に網羅検出が可能であることから、JAMSTECでは特許出願を行うと共に、科研費における新学術領域研究ネオウイルス学等を通して他機関との協力関係を構築し、医学・公衆衛生や農学分野での適用可能性を探る研究も開始しています。
[補足説明]
※1. ウイルスは自身で増殖することができず、他の生物細胞に寄生することでコピーを増やします。
※2.ウイルスはそのゲノムを構成する核酸の種類と複製様式により、6つのグループに大別されます。DNAをゲノムとするウイルスは一本鎖DNAウイルス、二本鎖DNAウイルス、そして、RNAをゲノムとするウイルスにはプラス鎖一本鎖RNAウイルス、マイナス鎖一本鎖RNAウイルス、二本鎖RNAウイルス、レトロウイルスがあります。レトロウイルスはreverse transcriving (RT)ウイルスとも呼ばれ、宿主細胞内でRNAからDNAに逆転写されることから、他のRNAウイルスと分けられています(図6)。
※3.二本鎖RNAウイルスはゲノムとして長鎖二本鎖RNAを有します。一方、一本鎖RNAウイルスではゲノム複製時に一時的に複製中間体として長鎖二本鎖RNAが形成されます。但し、レトロウイルス(RTウイルス)は逆転写されたDNAを介してゲノムが複製されるため、二本鎖RNAは形成されません(図7)。
※4.ウイルスの同定にはその遺伝子情報に基づく分類が不可欠です。一般に、既知ウイルスに対する解析ではPCR法が用いられますが、未知ウイルスを対象とする場合は、ゲノム解析が用いられ、DNAウイルスに対しては、微生物ゲノム解析と同様の簡便な手法が確立しています。一方、RNAウイルスについては、単離されたウイルスに対しては手間の掛かるゲノム解析手法は確立していますが、※5にも示す通り、複数種からなるウイルス集団を含む環境試料については、効率良い解析手法は確立していませんでした。
※5.多くの場合、RNAウイルスのゲノムは直鎖状の複数のゲノムセグメント(分節)からなり、その両末端は互いに類似した配列を有しています。従来法では部分的なゲノム情報を得ることしかできませんでしたが、今回開発したFLDSでは、末端配列を含む全長配列を得て(図8)、もとの完全なゲノムセットに再構築することが可能となりました(図9)。
このことは、従来法では難しいとされてきた未知遺伝子しか含まないセグメントを、ウイルス由来配列と識別できるようになったことも示しています。
※6. 採取した海水試料中を浮遊しているウイルス粒子を、その物理化学的性質に基づいて精製したもの。
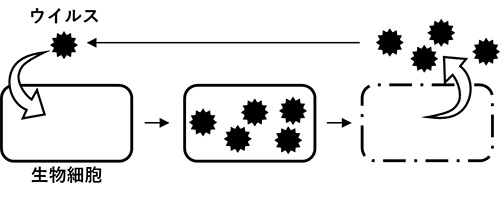
図1. 一般的にイメージされるウイルスの生活環。生物細胞へ感染したウイルスは細胞中で増殖して、細胞外に放出され、細胞はダメージを受ける。
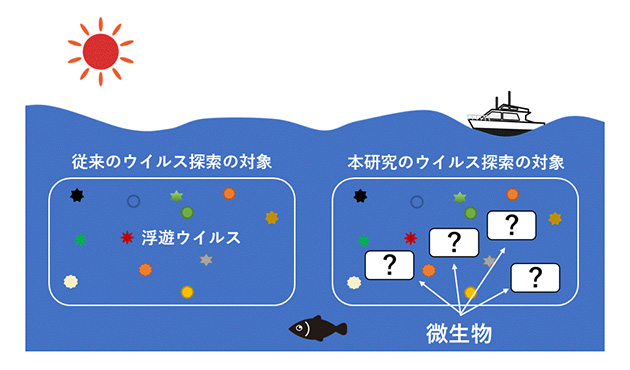
図2. 海洋における従来のウイルス探索対象(浮遊ウイルス)と、本研究のウイルス探索対象(浮遊ウイルス+微生物)。これまでのウイルス多様性解析においては、主に精製された浮遊ウイルス粒子(ウイルス粒子画分)のウイルス多様性が解析対象とされてきた。本研究では微生物細胞中(微生物細胞画分)とウイルス粒子画分におけるウイルス多様性を比較検討した。

図3. 本研究では、僅か合計10Lの海水に既知RNAウイルスグループの半分程度が存在することを示した。
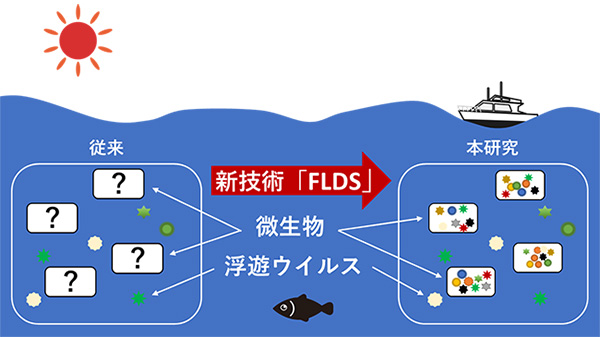
図4. 本研究により、特定のRNAウイルス群についてはそのほとんどが微生物細胞内で共存状態にあることが示唆された。
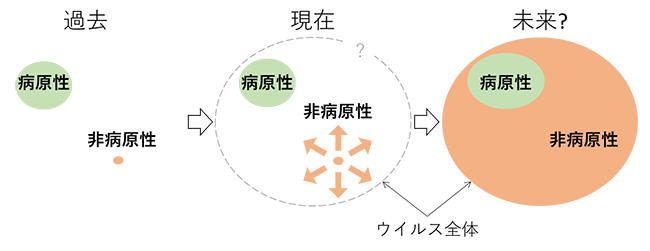
図5. 過去から現在のウイルス理解と、本研究により示唆される新たな“宿主と共存する”ウイルス像。従来知られるウイルスの殆どは宿主にダメージを与える病原性のものであった。本研究では、少なくともRNAウイルスにおいては、非病原性ウイルスの多様性が従来のウイルス像と比べ、遙かに大きなことを示した。今後の研究如何によっては、病原性を有すウイルスは極限られたものであることが示されるかもしれない。

図6. ウイルスのゲノム核酸種や複製様式による分類と、それぞれのグループに含まれる著名なウイルス
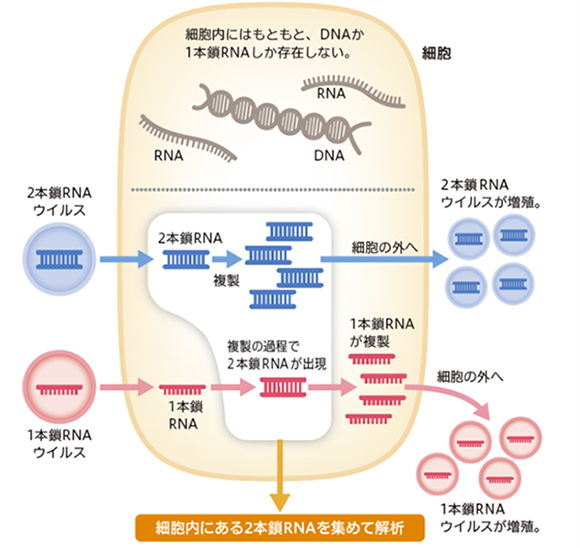
図7. 生物細胞の中に潜むRNAウイルスを効率的に探索するための原理。細胞内には多量の一本鎖RNAや二本鎖DNAが存在するため、そのままでは配列解読をしてもウイルスの配列がほとんど得られない。二本鎖RNAのみを解析対象とすることで、一本鎖RNAウイルス、二本鎖RNAウイルスの双方を特異的に検出することが可能となる。
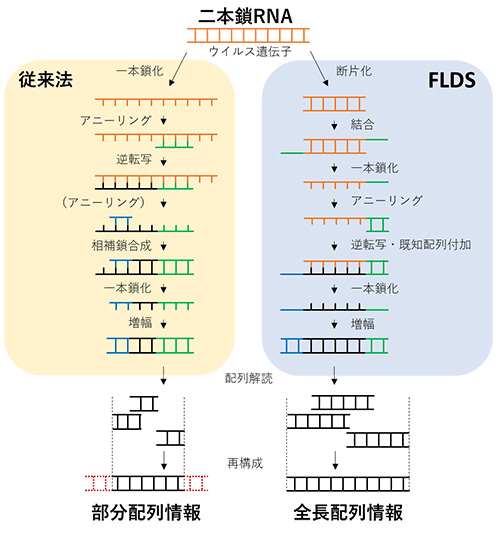
図8. 従来法およびFLDSにおけるcDNA合成原理と、得られるRNAウイルス配列情報の違い。従来法によるcDNA合成では元のRNA配列の末端部分が得られにくく、結果的に部分的な配列情報しか得ることができなかった。一方、FLDSでは断片化された二本鎖RNAの末端部分を含む全域がcDNAとなるため、全長配列情報を得ることが可能となった。オレンジ:RNA、黒:DNA、青・緑:cDNA合成に用いた配列。
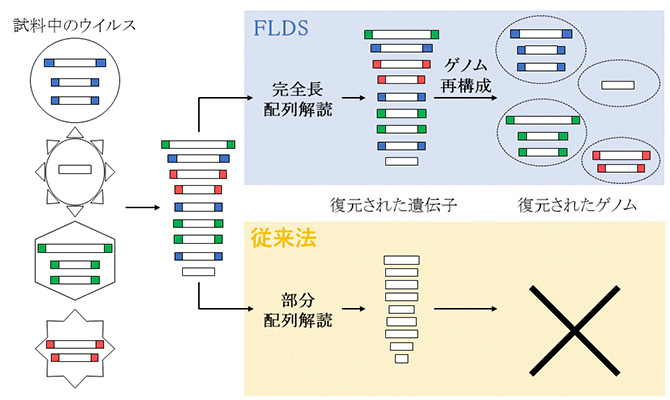
図9. FLDSでは、各ゲノム分節毎の末端配列の比較により、ウイルスゲノムの復元が可能であり、未知ウイルス遺伝子も検出可能である。従来法では末端配列の決定が困難であり、ウイルスゲノムの復元が困難であった。
- 国立研究開発法人海洋研究開発機構
(本研究について) - 海洋生命理工学研究開発センター 研究開発センター長代理 布浦 拓郎
- (報道担当)
- 広報部 報道課長 野口 剛