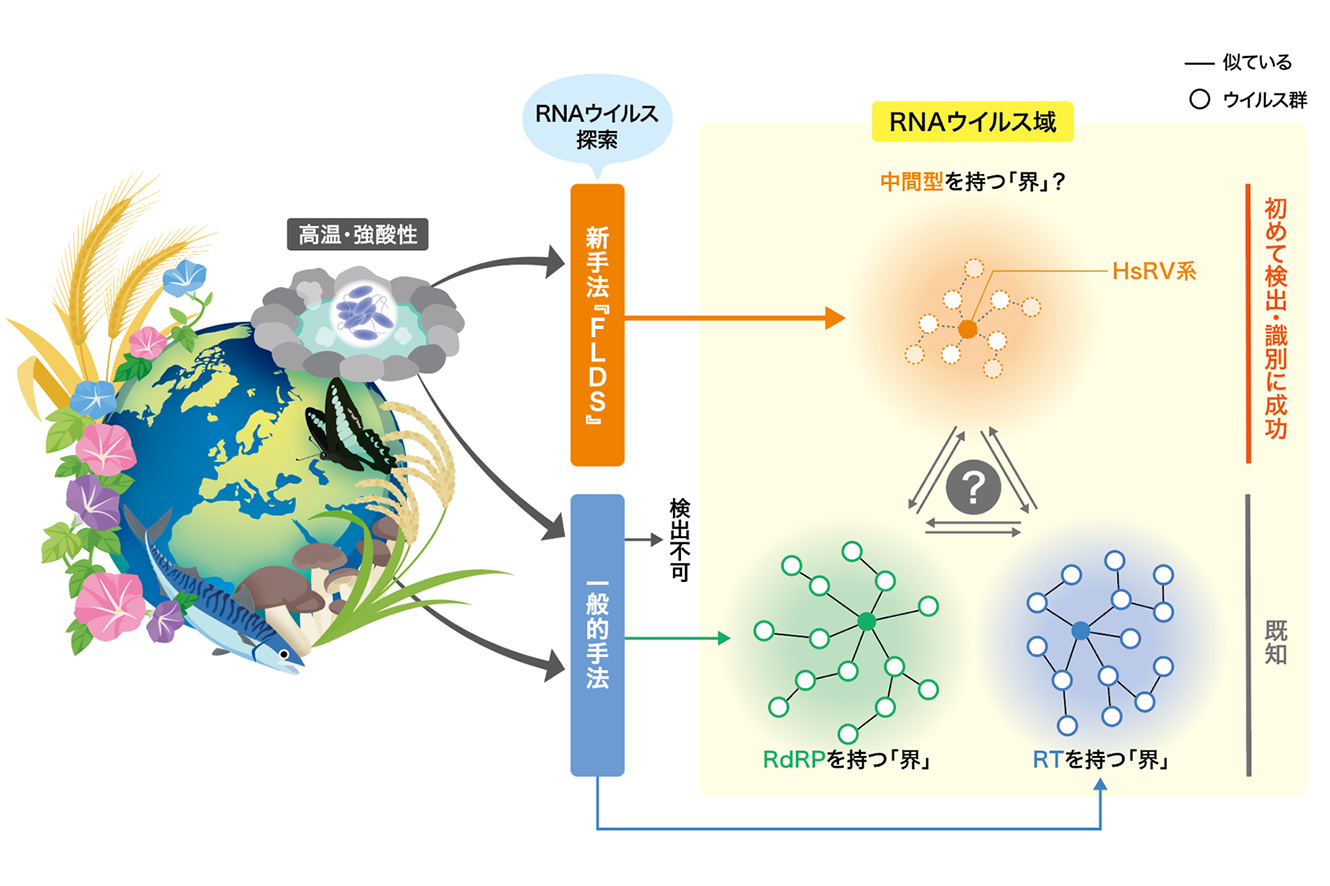
遺伝子による一般的なRNAウイルス探索手法では、既知の種と似たものを探すため、新規性の高い種は検出できないという難点があり、RNAウイルスの多様性の全容はまだ明らかになっていません。今回、本研究グループは、自ら2016年に報告した“新規性の高いRNAウイルス”も検出可能な新手法を用いて、高温酸性泉試料から新規のRNAウイルス系統を発見しました。現在RNAウイルスは自己複製酵素のタイプにより2つの「界」に分けられていますが、今回発見されたRNAウイルスはそのどちらの界とも異なる中間的な特徴を有しており、RNAウイルス第3の「界」となる可能性も示唆されています。さらに、今回見つかったウイルスの塩基配列を用いてデータベース検索をおこなったところ、近縁種は、高温酸性泉のような極限環境だけでなく、さまざまな水圏環境にも存在していることが分かりました。身近な環境においてすら見過ごされてきたRNAウイルス系統の発見は、RNAウイルス多様性の全容解明に向けた大きな一歩です。
ウイルスは特定の生物にしか感染できない
私たちが日常生活でウイルスという言葉を耳にするのは、ほぼ例外なく病気に関連するときなので、多くの人がウイルスに対してネガティブな印象を持っています。しかし実際は、ウイルスは途方もなく多様で、バクテリアから菌類、動植物などあらゆる生物に感染し、むしろヒトに感染する種は、ウイルス全体からみると無視できるほど低い割合です。
しかし、ウイルスはなぜそれほどまでに多様なのでしょうか?
その答えをひもとく鍵は「宿主特異性」にあります。生物はみな、進化の過程で、ウイルスや他の生物を含む周囲の環境と相互作用しながら、種ごとに異なる多様な細胞システムや抗ウイルス機構を獲得してきました。ウイルスもこれに対応する形で、宿主となるそれぞれの生物が持つ細胞システムや抗ウイルスの回避に特化した機構を獲得します。このような進化のイタチごっこの帰結として、どのウイルスも特定の生物にしか感染できません。これが宿主特異性と呼ばれる現象です。ウイルスはこの宿主特性を背景に、宿主となる生物の多様化に呼応する形で多様化を遂げ、現在のような驚くべき多様性を有するに至ったと考えられています。
環境中には膨大な量のウイルスが存在している
1989年、ノルウェー・ベルゲン大学の研究グループは透過型電子顕微鏡観察から、1mlの海水中に1000万以上のウイルス様粒子が存在していると報告しました。それまで見過ごされていたウイルスの膨大さ・多様性に光を当てたこの研究をきっかけに、海洋におけるウイルスの驚くべき量と多様性に注目が集まり、ウイルスの多様性の全貌を捉えようという取り組みが世界中で進められました。
まだ十分に探索されていない領域もあり、また、下述の通り、多くのウイルスは計数が困難であるため、諸説あるものの、地球上には1031オーダーという途方もない数のウイルスが存在しているという説が有力視されています。いずれにせよ、私たちの想像が及ばないほど多様かつ膨大なウイルスが地球上には存在していることが明らかになりつつあります。
未だ全容が見えないウイルスの多様性
ウイルスはゲノムを構成する核酸の種類と複製様式により、DNAをゲノムとするウイルスには1本鎖DNAウイルス、2本鎖DNAウイルス、そして、RNAをゲノムとするウイルスにはプラス鎖1本鎖RNAウイルス、マイナス鎖1本鎖RNAウイルス、2本鎖RNAウイルス、レトロウイルスの6つのグループに大別されています。
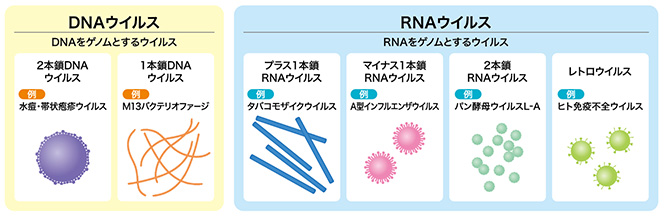
※プラス鎖1本鎖RNAウイルス、マイナス鎖1本鎖RNAウイルス:1本鎖RNAをゲノムとするRNAウイルスは、そのRNA分子が直接タンパク質合成に使われるのか、そのRNA分子を鋳型に合成される相補鎖のRNAがタンパク質合成のために使われるのかという違いにより、それぞれプラス鎖、マイナス鎖として分類されている。レトロウイルスはreverse transcribing (RT)ウイルスとも呼ばれ、宿主細胞内でRNAからDNAに逆転写されることから、他のRNAウイルスと区別されている。
比較的大きいゲノムを持つ2本鎖DNAウイルスであれば蛍光顕微鏡やフローサイトメータなどで粒子数をカウントできますが、1本鎖DNAウイルスやRNAウイルスの多くはゲノムが小さすぎて正確に計数することは困難です。さらに、ウイルスはさまざまな形態をもち、不定形のものや糸屑のような形のものなどもいることから、透過型電子顕微鏡などを使った観察でもすべてのウイルスを把握することはできません。
そこで近年、環境ゲノム解析を用いて環境中のウイルスゲノム配列を検出する研究が盛んに行われ、新規分類群の報告など、新たな知見が積み上がりつつあります。しかし、こうした研究は主にDNAウイルスを対象としており、RNAウイルスの多様性についての報告はDNAウイルスよりも少なく、その多様性の全容把握にはまだ程遠い状況です。私たちはRNAウイルス検出のための新たな技術を駆使して、その全貌に迫りたいと考えています。
見過ごされてきた「持続型」ウイルス
手法的な問題だけでなく、動植物の疾病や微生物の死滅など、宿主に大きなダメージを与えるウイルスが注目されやすかったという背景も、本来のウイルスの多様性を認識する障壁となってきました。
今回、私たちが研究対象としたRNAウイルスは生活型の違いにより、「急性型」と「持続型」に分けられます。言葉の響きから医学分野で言われる急性期や潜伏期などを連想されるかもしれませんが、宿主側の病気の進行とは全く関連のないウイルス学用語です。
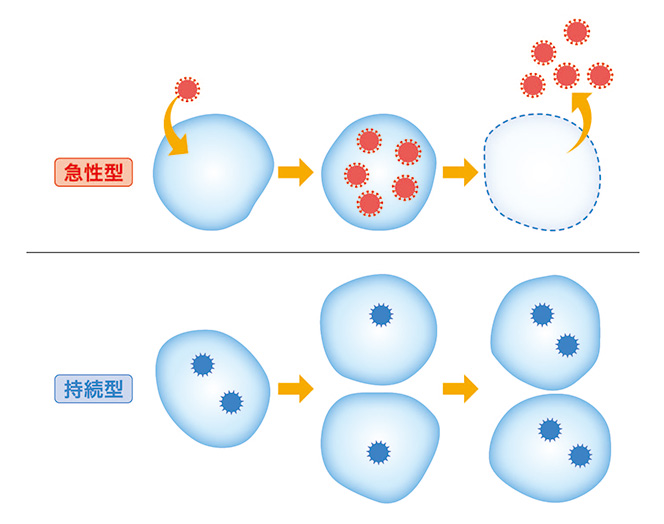
急性型とは、宿主の細胞に外部から侵入し、細胞の外部に脱出し、主に細胞外に出たウイルス粒子を介して他の細胞へ伝播していく生活型を意味します。急性型ウイルスは、細胞から脱出する際に、細胞溶解に代表されるような「ダメージ」を引き起こすことがあります。病原ウイルスとしてよく知られる、インフルエンザウイルスやコロナウイルスなどがこれにあたります。
しかし、急性型でも、動植物や微生物の死や病気につながるほど大きなダメージを起こすケースはまれで、多くの急性型ウイルスは、生物個体や細胞に大きなダメージを与えない程度の緩やかなレベルのウイルス粒子の生産によって世代を繋いでいると考えられます。つまり、穏やかではない名前にそぐわず、ほとんどの急性型ウイルスは明瞭な病原性を持っていないのです。
それに対して、持続型ウイルスは細胞溶解を伴わず、主要な伝播が宿主の細胞分裂によって行われます。持続型ウイルスは宿主細胞への侵入および細胞からの脱出能力を欠いているため、ほとんどの種が宿主と共生的な関係にあるのではないかと考えられています。身近な例としては、イネやピーマンなどの細胞にも持続型RNAウイルスが潜んでいます。そうしたウイルスは、私たちが普段口にしている健全なイネやピーマンなどにも普通に含まれています。
持続型RNAウイルスや病原性を持たない急性型RNAウイルスは私たちの目に止まる機会がこれまでほとんどなかったので、その大半が見過ごされてきました。しかし、最近の研究から、地球上のどこにでも存在することが分かってきています。定量的な比較はまだできていませんが、私たちのこれまでの研究から、真核微生物を宿主とするRNAウイルスの多くが持続型である可能性が高いことが示されています。
原核生物であるバクテリアに感染するRNAウイルスも、従来はバクテリア細胞の死滅を指標に探索されてきましたが、この方法では、急性型の中でもダメージが大きな型しか見つけられませんでした。実際、この方法でこれまでに得られたバクテリアに感染するRNAウイルスの種類は限られていました。しかし、近年、RNAウイルスのゲノムについても、網羅的に環境ゲノムを探索する研究が進んでおり、その結果、バクテリアを宿主とするRNAウイルスも極めて多様であることが示されつつあります。
RNAウイルスゲノムを網羅的に解析できるFLDS法
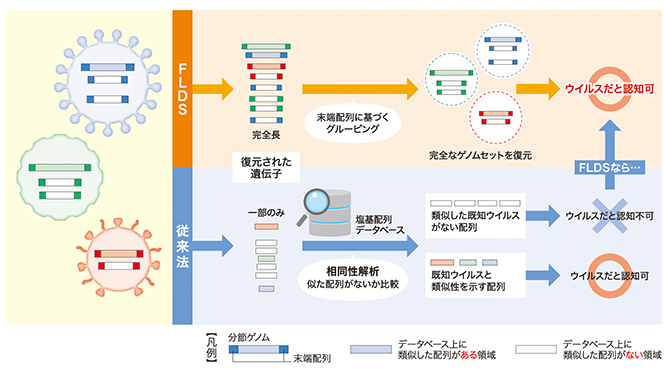
現在主流となっているRNAウイルスの探索手法は、宿主と想定される細胞から得たメッセンジャーRNA(mRNA)をランダムに解読し、その中に既知のRNAウイルスの配列と似たものを探すという方法です。そのため、すでに報告されているRNAウイルスの配列と似ていない新規性の高いRNAウイルスは検出できないという限界があります。
本研究グループが2016年に発表した「FLDS法」は、この限界を一部突破できるRNAウイルス検出手法です。
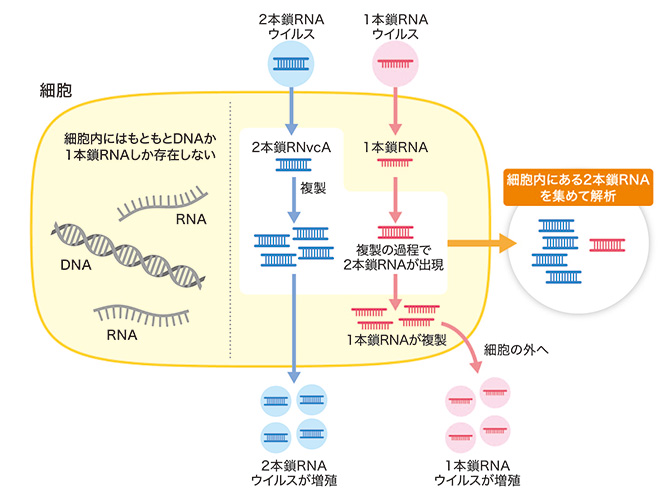
FLDS法では、宿主細胞中に存在するRNAウイルス由来の長鎖2本鎖RNAに着目します。長鎖2本鎖RNAはRNAウイルスに感染していない細胞にはほとんど存在しませんが、RNAウイルスに感染するとそのゲノムやゲノムの複製中間体としてウイルス由来の長鎖2本鎖RNAが細胞内に蓄積されます。これを利用して、2本鎖RNAのみを解析対象とすることで、1本鎖RNAウイルス、2本鎖RNAウイルスに由来する配列を特異的に検出することが可能となります。
また、RNAウイルスの多くは複数の分節ゲノムから構成されたゲノムセットを持ちますが、FLDS法では、これらの分節ゲノムの末端配列がウイルス種ごとに高い類似性を示すことを利用して、種ごとの完全なゲノムセットを復元することができます。これにより、これまでの探索手法では、実際にはRNAウイルスに由来する遺伝子配列であっても、既知のウイルスと類似配列がない場合は検出できませんでしたが、FLDS法ではウイルス配列として検出することができます。
*プレスリリース:海洋微生物の中に隠された新しいウイルスワールドを発見~RNAウイルス網羅検出技術の開発と海洋微生物への適用~(2018年9月7日)
極限環境から未知のRNAウイルス系統を発見。第3の「界」の可能性も
本研究では、FLDS法を用いて、過去の解析ではRNAウイルスが見つかっていない高温(>70℃)かつ酸性(<pH2)の温泉から採取した微生物試料を解析しました。
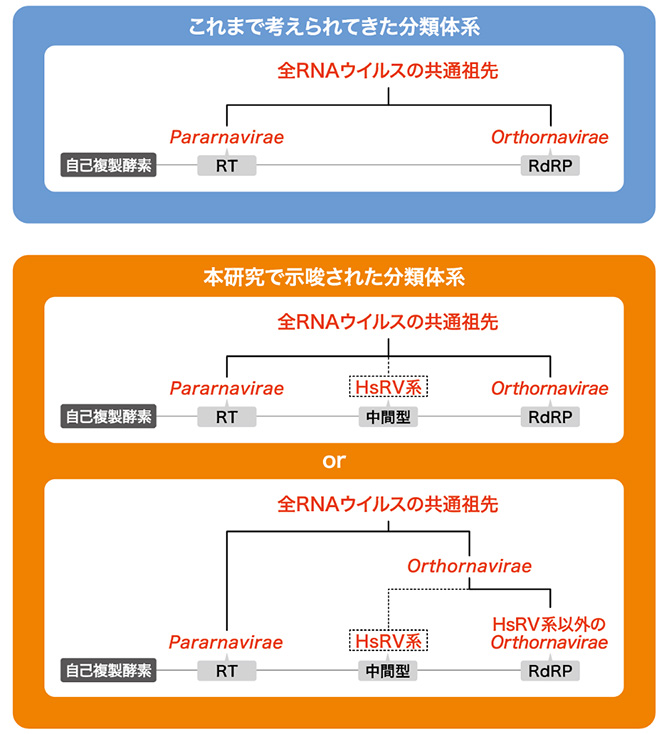
その結果、これまでのRNAウイルスの分類体系に属さない新規分類群を見出しました。私たちは、採取された環境にちなんで、この新規RNAウイルス系統を「hot spring riboviruses(HsRV)」と名付けました。
これまでRNAウイルスは、自己複製酵素としてRT (reverse transcriptases: 逆転写酵素)を持つレトロウイルス(Pararnavirae界)とRdRP(RNA-dependent RNA polymerases:RNA依存性RNAポリメラーゼ)を持つ非レトロRNAウイルス(Orthornavirae界)という二つの「界」(※)に分けられてきました。本研究以前に遺伝子レベルで見出された数十万種ものRNAウイルスはすべてこのどちらかの界に属していました。RTとRdRPは異なる酵素ですが、類似の構造を持つことから、共通の起源を持つと考えられています。 驚くべきことに、今回発見されたHsRVから予測された自己複製酵素の構造は、すでに知られたRdRPともRTとも異なる特徴を有していました。このことから、新しい門(Artimaviricota*を提唱)に該当することは間違いなく、新たな界となる可能性すらあります。さらに、HsRVの自己複製酵素の予測構造はRTとRdRPの中間的な特徴を有していたことから、RTとRdRPを繋ぐミッシングリンクを埋める存在として、今後さらに研究を進めることで、RNAウイルスの進化に関する新たな情報が得られると期待されます。
*逆転写酵素RT[発音記号əːrtíː]の韻と、リトアニア語で「近い」を意味するartimaを掛けて、RTの隣を含意して命名。
※ウイルスは上位のものから域・界・門・綱・目・科・属・種の分類階級に分けられる。今回見つかったHsRVの分類階級はまだ定まっていないが新たな「界」となる可能性があり、私たち人間が認知できるウイルスの多様性の窓を大きく開く成果と言える。
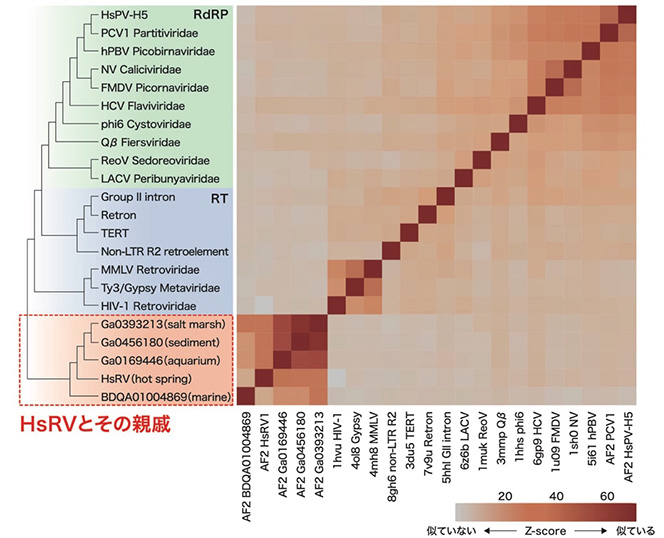
HsRVは極限環境以外にも存在
これまでに報告されているRNAウイルス配列に似ていない配列を持ったウイルス種を検出できたのは、FLDS法を用いたからこその成果です。実際、今回FLDS法でHsRVが検出された同一試料に対して、既存のRNAウイルスゲノムデータベースを用いた一般的なRNAウイルス遺伝子探索を実施したところ、HsRVの存在は認知できませんでした。
また、HsRVを同定したことで、これまでRNAウイルスとは認識されていなかった遺伝子配列が、HsRVに近縁なRNAウイルスゲノムとして識別できるようになったことも、本研究の大きな成果です。実際、塩基配列データベースでHsRVに近縁な配列を検索したところ、今回HsRVを発見したような極限環境とは異なる普通の水圏環境にもHsRV近縁種が存在することが示唆されました。HsRVがどれほど多様なRNAウイルスで構成されているグループであるのか、今後さらなる解析が必要です。また、現段階ではHsRVに属する種が少なく、系統解析の精度が不十分であるため、HsRVが門に相当するのか、界に相当するのか、明確な答えを出すことができませんが、HsRVに属する種数が増えることで、系統解析の信頼性も向上し、ふさわしい分類階級が定まると予想されます。
なお、HsRVは系統的に類似した既知種が存在しないため、急性型か持続型かは不明ですが、ヒトに関連するデータベースとの照合の結果、HsRVの近縁種は見つからなかったことから、HsRVの近縁種がヒトに対して病原性を示す可能性は極めて低いと言えます。宿主は明確にはわかっていませんが、採取された環境などから、生命の共通祖先に近い高度好熱性のバクテリアの可能性が高いと考えられます。
「未知のウイルス群発見」のその先へ

自然界には、今回見つかったHsRVや、その宿主の可能性が高いと考えられる好熱好酸性バクテリアなど、私たちの意識にも上らないようなちっぽけな、しかし無数のウイルスや微生物が存在しています。そうした微生物をはじめとする多種多様な生物の働きによって、それぞれの地域の生態系、ひいては地球上の生物多様性が維持されています。私たちの身の周りにも、まだまだ人間が認知できていない種や分類群、そして生物の生態や進化に関わる未知の因子が無数に存在しています。RNAウイルスはそういった存在の代表と言えるでしょう。
地球上にはこれまでの認識をはるかに上回る多様かつ膨大な数のRNAウイルスが存在していることがわかりつつあります。病原性を示さない場合でも、RNAウイルス感染は宿主細胞の機能に一定の影響を与えることから、個々の生物のふるまいだけでなく、無数の生物同士が相互につながりあった地球生態系を理解する上で、こういった目には見えない微生物・ウイルスの存在をまずは認知することが重要です。本研究はその第一歩になる重要な成果と言えます。
イラストレーション:いさか なおこ